Пикософт
Подписаться на RSSОбоснование 3Д Генетического кода
https://www.nature.com/articles/s41467-022-30390-9
Кодон-специфические графики Рамачандрана показывают, что конформация главной белковой цепи зависит от кодона
Рассылки Лаборатории Наномир, Выпуск 1323
(ГУГЛ знает)
У нас есть одна таблица - 3D генетический код и 1 таблица - результаты корреляционного анализа по 300+ белкам. Для "каких попало" белков средняя корреляция ~96%, для мембранных белков, представляющих собой длинные спиральные участки, корреляция 100%.Структура статьи-сообщения предельно проста.
Целью исследования была проверка гипотезы синонимичности генетического кода.Нулевая гипотеза: Если генетический код (2D код) синонимичен, то глицин в любой спирали (альфа, пи, бета, 310) будет встречаться закодированным любым из кодов: GGA, GGC, GGG, GGT.Проверка показывает, что эта гипотеза ошибочна. В альфа спирали глицин кодируется кодом GGC, в пи-спирали GGT, в 310-спирали GGG, в бета-спирали GGA.Таким же способом составляется вся таблица, т.е. для всех аминокислот.
Рис. 2 Различные предпочтения к образованию вторичных структур синонимичными кодонами проявляются в распределении двугранных углов. Из двух кодонов GTA и GTT, транслирующих валин, GTA имеет на 8 % меньшее предпочтение к β-моде и на 9,4 % большее - к α-моде. Предпочтения проявляются в относительных весах соответствующих мод на диаграмме Рамачандрана, что видно на представленных здесь предельных распределениях двугранного угла φ. Если добавить ограничение на вторичную структуру, то распределения двух синонимичных кодонов становятся неразличимыми. Благодаря учету вторичной структуры наш анализ становится устойчивым к различиям в распределении, возникающим из-за различий в предпочтениях. Показаны оценки плотности ядра, заштрихованные области обозначают 10-90% доверительного интервала, рассчитанных на 1000 случайных бутстрапов.
Кушелев: Проще говоря замена кодона при определённых условиях влияет на структуру белка. В случае синонимичности кодонов это невозможно. Не будем вдаваться в тонкости изощрённых методов, с помощью которых авторы статьи получили этот надёжный и убедительный результат. Обратим внимание лишь на сложность получения этого результата на основе данных рентгеноструктурного анализа, который, например, не отличает альфа-спираль от пи-спирали и от фрактальной спирали Кушелева. Из-за этого трудно заметить влияние кодонов, формирующих 1/4 альфа-спирали от влияния кодонов, формирующих 1/5 пи-спирали. Ведь рентгеноструктурный анализ их не различает. Более того, многие вторичные структуры, состоящие из участков альфа-спиралей, специалисты по РСА считают состоящими из бета-цепей. В связи с этим банк белковых структур (PDB) "зашумлён" систематическими ошибками. Тем не менее, мощные методы исследования помогли авторам обнаружить зависимость структуры главной белковой цепи от замены кодонов на "синонимы". Теперь мы можем смело ссылаться из статьи "3D генетический код" на книгу "Пикотехнология белков", т.к. существование 3D генетического кода надёжно подтверждено опровержением гипотезы синонимичности кодонов. А это значит, что нет необходимости срочно писать всю серию статей от "Кольцегранные модели молекул" и далее. Можно писать сразу статью "3D генетический код" со ссылками на книгу "Пикотехнология белков", в которой опубликована таблица 3D генетического кода. Обоснование 3D генетического кода, как мы видим, уже есть, а проверку конкретной таблицы мы опубликуем в статье в виде результатов корреляционного анализа.
Проверка корреляции подтверждает наличие 3D генетического кода (см. вложенный файл)
Модели и эксперименты
В таблице 3D генетического кода каждому триплету кодирующей нуклеотидной последовательности соответствует не только аминокислота, но и вариант её установки в растущую структуру белка. Например, триплет GGC кодирует глицин в составе альфа-спирали, GGA – глицин в составе бета-спирали, GGG – глицин в составе 310-спирали, GGT – глицин в составе пи-спирали.
Файл Fasta и 2D диаграмма Пикотех
Файл Fasta и 2D диаграмма Пикотех
Альфа-фолд знает про сверхдлинные альфа-спирали!
Существующие "мокрые" методы анализа белковых структур обычно не отличают даже трёхгранную призму (фрактальную 75-спираль Кушелева) от других типов вторичной структуры (альфа-, пи-, 310-спиралей).
Сверхдлинные 310-спирали, как мы видим, тоже современные методы структурного анализа обнаружить обычно не могут. Хотя Альфа-фолд уже нарисовал длинные спиральные участки. 310-спираль для современных методов обычно неотличима от пи-спирали и от альфа-спирали.
Это снова сверхдлинная фрактальная 75-спираль Кушелева (бета-призма по официальной классификации).
На этот раз Альфа-фолд предсказывает множество спиральных участков.
Это значит похожие белки исследовались с помощью РСА/ЯМР и т.д. и были приняты за спираль.
Это и есть спираль, но фрактальная, состоящая из чередующихся участков n*(1/4 витка альфа - 1/5 витка пи -)
Примеры фрактальных спиралей
Фрактальная спираль - спираль, скрученная из спирали.
Пространственная структура до фолдинга третичной структуры - правильная вторичная структура в пространстве ( ~ 20 000 АО).
Наконец, удалось добраться до прямых результатов РСА для фрактальной спирали!
https://i.ibb.co/CWZT7z7/Q96JA1.png
https://www.uniprot.org/uniprotkb/Q96JA1/entry
Речь идёт о внедрении готовых технологий. Я уже сегодня могу выполнять заказы, например, по определению структур транс-мембранных белков. Известно, что они спиральные, но конкретизировать тип спирали РСА/ЯМР не могут. А программа Пикотех показывает конкретный тип спирали. Это может быть альфа-спираль (показана красным цветом). Может быть 310-спираль (показана оранжевым), может быть пи-спираль (синяя), может быть бета-спираль (зелёная), может быть метиониновая спираль (сиреневая), а может быть фрактальная спираль, например, 75-спираль Кушелева. Она изображается чередованием цветов "розовый-голубой". Другие фрактальные спирали тоже "полосатые", а типов фрактальных спиралей очень много. И все они на 100% правильно показываются по таблице 3D генетического кода. Нетрудно догадаться, что после публикации серии статей появится лавина заказов на структуры белков, тем более, что бесплатных примеров опубликовано в рассылке более 200 000. Это даже больше, чем вся база белковых структур (PDB), которая насчитывает около 150 000 структур. А в рассылке опубликованы около 110 000 структур только белков человека. В каждом выпуске рассылки, где опубликованы очередные 200 белковых структур, я фактически дарю человечеству бесценную информацию о структурах этих белков. Ведь подавляющее большинство белков (около 97%) не кристаллизуются и поэтому недоступны для РСА. Их не могут определить ни за какие деньги. А я эти структуры дарю сотнями.
Публикации
Программа Пикотех помогает биологам делать научные открытия
Монография, статьи
ЧИТАТЬ ОНЛАЙН Монография посвящена 3D-структуре молекул и полимеров живых систем. Дан анализ современного понимания таких фундаментальных понятий, как физический объём атома, химическая связь, генетический код. На основе статистического анализа экспериментальных данных о структуре белка обосновано кодирование его вторичной структуры и структурного полипептидного шаблона в геноме. Предложена дополненная таблица генетического кода белков и пептидов, которая легла в основу геометрического алгоритма программ декодирования структурного шаблона белка Molecular Constructor и Picotech. Сформулирована гипотеза о перекодировании информации третьего нуклеотида кодона в соответствующий ротамер пептидной связи непосредственно 3D-структурой изоакцепторной тРНК. Математический анализ сопряженности значений углов φ и ψ (карта Рамачандрана) выявил периодичность их изменения, что позволило обосновать механизм посттрансляционного фолдинга белка.
Книга рассчитана на специалистов, занимающихся исследованиями в области молекулярной биологии, биоинформатики, биохимии, биофизики.
Табл.: 36 . Ил.:117 . Библиогр.: 227 назв.
2. Кушелев А.Ю., Соколик В.В. Пикотехнология – новый подход в моделировании пространственной структуры белка / Заочная Международная научно-практическая конференция «Современная наука: тенденции развития» (24 января 2012), Краснодар: НИЦ Априори. – 2012. – С.203-207.
5. Kushelev A.Y., Pisarzhevsky S.A. The construction of the scale model of the spatial structure of the protein on its nucleotide sequence. // "Молекулярная биология" (не опубликована).
6. Кожевников Д.Н. Кольцегранные модели молекул. Журн. физ. химии. - 1996. - Т. 70. - No 6. - С. 1134-1137.
7. Кожевников Д.Н. Использование моделирования в обучении в контексте понимания и усвоения категории сложности. // Вестник Московского университета. Серия 20. Педагогическое образование. - 2015, N3 (июль-сентябрь). - с.21-34. -
8. Назарова Т.С., Тихомирова К.М., Кудина И.Ю., Кожевников Д.Н. Теоретические основы стандарта учебно-материальной базы общего среднего образования. (Федер.гос. науч. учреждение "Ин-т содерж. и методов обучения" РАО). - М.: Нестор-История, 2014. - 168с.
9. Кожевников Д.Н. Криволинейные контурные конструкторы "Крикоко" и "Тайкон". Методические рекомендации по использованию. - м.: МГДД(ЮТ)Т, 2013. - 72с.
10. КОЖЕВНИКОВ Дмитрий Николаевич. Создание и использование комплекса моделей атомов и молекул для изучения строения вещества в курсе химии средней школы. 13.00.02 – теория и методика обучения и воспитания (химии в общеобразовательной школе) (по педагогическим наукам). Диссертация на соискание ученой степени кандидата педагогических наук. Научный руководитель: член-корреспондент РАО, доктор педагогических наук, профессор Назарова Т. С. Москва - 2004
11. V.V.Sokolik. “Implementation of the 3D genetic code of proteins is isoacceptor tRNAs”, Journal Medical and Clinical Chemistry. 2019.V. 21, No. 3 (Appendix). P. 46-47. ISSN 2410-681X)
12. В. Соколик. СТРУКТУРНЫЕ ПРЕДПОСЫЛКИКИ АГРЕГАЦИИ БЕТА-АМИЛОИДНОГО ПЕПТИДА Украiнський вiсник психоневрологii – 2009. – Т.46, вип.8.- С. 116—121.
13. Sokolik. Modeling of the 3D structure of apoliprotezin E3 under its determining nucleotide sequence. III International Conference "Actual problems of biology, nanotechnology and medicine". Theses of reports (October 1-4, 2009), Rostov-on-Don. - P. 54.
15. В.В. Соколик "Никакой дополнительной информации, большей, чем та, что содержится в ДНК, для сворачивания белка не требуется" Матеріали ХІ Укріїнського біохімічного конгресу (06-10 жовтня 2014), The UkrainianBiochemical Journal, 2014, V. 86, №5 (Supplement 1), P. 37-38.
16. Соколик В.В. Пространственная структура гомологов основного актина и бета-актина 1 различна. - Материалы I Международной научно-практической конференции "Наука и современность - 2010" в 3-х частях (Под общ. ред. С.С.Чернова). Часть 1. - Новосибирск: Сибпринт, 2010. - с.41-46(278с.).
17. В.В. Соколик "Никакой дополнительной информации, большей, чем та, что содержится в ДНК, для сворачивания белка не требуется" Матеріали ХІ Укріїнського біохімічного конгресу (06-10 жовтня 2014), The Ukrainian Biochemical Journal, 2014, V. 86, №5 (Supplement 1), P. 37-38.
18. Соколик В.В. Кодирование вторичной структуры и структурного шаблона белка в геноме эукариот / НАУЧНЫЙ ФОНД "БИОЛОГ", Ежемесячный научный журнал. – 2014. – № 3. – С. 73-76.
19. Соколик В.В. Никакой дополнительной информации, большей, чем та, что содержится в ДНК, для сворачивания белка не требуется / Ukr. Biochem. J., 2014. – 86, 5 (Suppl. 1) Матеріали ХІ Укріїнського біохімічного конгресу (06-10 жовтня 2014), Київ, c. 37-38
20. Соколик В.В. Предсказание пространственной структуры белка insilico на основе информации генома и геометрического алгоритма – альтернатива квантово-механическому подходу // Материалы Международной научной конференции «Математическое и компьютерное моделирование в биологии и химии. Перспективыразвития» (28-30 мая 2012), Казань. – 2012. – С.155-158.
21. Sokolik V.V. Protein is coded in genome and synthesized in ribosomes as a structural template of a rotameric version sequence of peptide bound configuration // The International Moscow Conference on Computational Molecular Biology, МССМВ-11, Moscow. – 2011. – P. 347–348.
22. Sokolik V.V. Algorithm of protein structural template decoding according to its determined nucleotide sequence // Fist International Conference “Fundamental medicine: From scalpel toward Genome, Proteome and Lipidome”, Pax Grid Virtual Conferences, Kazan. – 2011. – P. 117–119.
23. Sokolik V.V. Modeling of the individual structural template of protein on determining it nucleotide sequences // VII Международнаяконференцияпобиоинформатике, регуляцииструктурыгеномовисистемнойбиологии. BGRS\SB-2010, Новосибирск. – 2010. – С. 275.
24. Соколик В.В. Способ моделирования пространственной структуры белка по детерминирующей его нуклеотидной последовательности // Биофизический вестник. – 2010. – Вып. 24 (1). – С. 31-45.
25. Соколик В.В. Геометрия аминокислот / Материалы XV Международной научно-практической конференции «Наука и современность – 2012» (НС-15), (14 марта 2012), Новосибирск. – 2012. – С. 13-18.
26. Соколик В.В. Загадка изоакцепторных тРНК / Материалы II Всероссийской Интернет-Конференции «Актуальные проблемы биохимии и бионанотехнологии» (15-18 ноября 2011), Казань, Россия. – С. 11-15.
27. Соколик В.В. Кодирование торсионного угла ω пептидной связи в белке / IV Международная конфер. "Актуальные проблемы биологии, нанотехнологий и медицины". (22-25 сентября 2011), Ростов-на-Дону. – С. 60-61.
28. Соколик В.В. Карта Рамачандрана: ротамерия пептидной связи и фолдинг белка / Материалы VII Международной научно-технической конференции «Актуальні питання біологічної фізики і хімії». Тезисы докладов БФФХ-2011 (26-30 апреля 2011 г.), Севастополь.– С.137-139.
29. Соколик В.В. Ротамерные варианты конфигурации пептидной связи и их кодирование в геноме / Матеріали X Українського біохімічного з’їзду. Тези доповідей (13—17 вересня 2010 р.), Одеса. – С. 105-106.
30. Соколик В.В. Пространственная структура гомологов основного актина и α-актина 1 различна / Сборник материалов I Международной научно-практической конференции «Наука и современность – 2010» в 3-х частях / Под общ. ред. С.С. Чернова - Новосибирск: «СИБПРИНТ», 2010. – 278 с. Ч. 1, С. 41-46.
32. Соколик В.В. Моделирование пространственной структуры белка по детерминирующей его нуклеотидной последовательности / Материалы VI Международной научно-технической конференции «Актуальні питання теоретичної і прикладної біофізики, фізики і хімії». Тезисы докладов БФФХ-2010 (26-30 апреля 2010 г.), T.1, Севастополь.– С.201-204.
32. Cоколик В.В. Кодирование вторичной структуры и структурного шаблона белка в геноме эукариот / НАУЧНЫЙ ФОНД "БИОЛОГ", Ежемесячный научный журнал. – 2014. – № 3. – С. 73-76.
33. Соколик В.В. Никакой дополнительной информации, большей, чем та, что содержится в ДНК, для сворачивания белка не требуется / Ukr. Biochem. J., 2014. – 86, 5 (Suppl. 1) Матеріали ХІ Укріїнського біохімічного конгресу (06-10 жовтня 2014), Київ, c. 37-38
34. Соколик В.В. Предсказание пространственной структуры белка insilico на основе информации генома и геометрического алгоритма – альтернатива квантово-механическому подходу // Материалы Международной научной конференции «Математическое и компьютерное моделирование в биологии и химии. Перспективыразвития» (28-30 мая 2012), Казань. – 2012. – С.155-158.
35. Sokolik V.V. Algorithm of protein structural template decoding according to its determined nucleotide sequence // Fist International Conference “Fundamental medicine: From scalpel toward Genome, Proteome and Lipidome”, Pax Grid Virtual Conferences, Kazan. – 2011. – P. 117–119.
36. Sokolik V.V. Modeling of the individual structural template of protein on determining it nucleotide sequences // VII Международнаяконференцияпобиоинформатике, регуляцииструктурыгеномовисистемнойбиологии. BGRS\SB-2010, Новосибирск. – 2010. – С. 275.
37. Соколик В.В. Способ моделирования пространственной структуры белка по детерминирующей его нуклеотидной последовательности // Биофизический вестник. – 2010. – Вып. 24 (1). – С. 31-45.
38. Соколик В.В. Геометрия аминокислот / Материалы XV Международной научно-практической конференции «Наука и современность – 2012» (НС-15), (14 марта 2012), Новосибирск. – 2012. – С. 13-18.
39. Соколик В.В. Загадка изоакцепторных тРНК / Материалы II Всероссийской Интернет-Конференции «Актуальные проблемы биохимии и бионанотехнологии» (15-18 ноября 2011), Казань, Россия. – С. 11-15.
40. Соколик В.В. Кодирование торсионного угла ω пептидной связи в белке / IV Международная конфер. "Актуальные проблемы биологии, нанотехнологий и медицины". (22-25 сентября 2011), Ростов-на-Дону. – С. 60-61.
41. Соколик В.В. Карта Рамачандрана: ротамерия пептидной связи и фолдинг белка / Материалы VII Международной научно-технической конференции «Актуальні питання біологічної фізики і хімії». Тезисы докладов БФФХ-2011 (26-30 апреля 2011 г.), Севастополь.– С.137-139.
42. Соколик В.В. Ротамерные варианты конфигурации пептидной связи и их кодирование в геноме / Матеріали X Українського біохімічного з’їзду. Тези доповідей (13—17 вересня 2010 р.), Одеса. – С. 105-106.
43. Соколик В.В. Пространственная структура гомологов основного актина и α-актина 1 различна / Сборник материалов I Международной научно-практической конференции «Наука и современность – 2010» в 3-х частях / Под общ. ред. С.С. Чернова - Новосибирск: «СИБПРИНТ», 2010. – 278 с. Ч. 1, С. 41-46.
2D диаграммы трехмерной формы белков
Шедевры белковой архтектуры
Высоко периодичные структуры белков
ПРОГРАММИРУЕМ ДЕКОДЕР кода белка в 3D структуру
КРАТКАЯ 2D диаграмма ПИКОТЕХ
Красный - альфа-спираль.
Оранжевый - 310-спираль.
Синий - пи-спираль.
Розовый - одиночный код альфа/310 спиралей.
Бирюзовый - одиночный код пи-спирали.
Тёмно-зеленый - бета-спираль.
Зелёный - одиночный код бета-спирали
Сиреневый - метиониновая спираль. У неё более крупный шаг "резьбы", чем у обычной альфа-спирали.
Жёлтый - сустав пролина
Черный в сокращённом представлении и белый в развернутом означают либо неизвестный код, либо конец трансляции.
Циклическое повторение цветов (композиционных кодов) - программная спираль.
Программные спирали - повторение последовательности композиций. Например, один код альфа-спирали, затем один код пи-спирали. n(35) задаёт программную спираль, а n3 или n5 - простые спирали (пи-спираль и альфа-310-спираль).
232323 - программная n(23)-спираль
141414 - программная n(14)-спираль
Фрактальная 585557-спираль с периодом 6.
Фрактальная 1114477-спираль с периодом 7.
Маленький белок с длинной альфа-спиралью (красный).
Переходы фрактальных спиралей разных типов.
Фрактальная 33333555755887775-спираль с периодом 17
Фрактальная 8755755877P-спираль с периодом 11.
Этот маленький белок состоит из нескольких периодов фрактальной спирали, состоящей из альфа-спиральных участков (красный), 310-спиральных участков (оранжевый) и пи-спиральных участков (синий).
Фрактальная 3333333P77P77P77875-спираль с периодом 19.
Фрактальная квази-спираль с типовым периодом 7.
Фрактальная 33333335587877558-спираль с периодом 17.
Фрактальная квазиспираль с типовым периодом 19.
Фрактальная спираль с дробным периодом (~14.7) переходит в 411617P75755577-спираль с периодом 15.
Фазированный фрактал (квази-спираль)
Фрактальная 3333311114885575-спираль с периодом 16.
Фрактальная спираль с периодом 32.
Фрактальная спираль с периодом 21.
Фрактальная 111181111757575577-спираль с периодом 18.
Фрактальная спираль с периодом 29.
Фрактальная спираль с периодом 76.
Фрактальная Q-спираль (75-спираль Кушелева) переходит в бета-спираль (L-спираль).
Фрактальная квазиспираль в стиле куфи.
Фрактальная 5557555P75577-спираль с периодом 13.
Фрактальная квазиспираль с типовым периодом 19.
Три поколения алгоритмических конструкторов белков.
Программируем декодер кода белка в 3D структуру
Модель фрактальной спирали из суставно-стержневого конструктора.

Это - фрагмент фрактальной спирали, состоящий из двух прямых участков пи-спирали (по 10 аминокислотных остатков), соединённых прямым альфа-спиральным усастком.

Вид вдоль оси одного из пи-спиральных участков.

Красной трубочкой отмечена единственная водородная связь, стабилизирующая альфа-спиральный участок, т.е. соединяющая его первый остаток с 4-ым.

Связи С-Сальфа показаны белыми трубочками. На участках пи-спирали эти трубочки с голубыми и зелеными полосками, а на участке альфа-спирали эти трубочки с красными и желтыми полосками.
Модель из суставно-стержневого конструктора показала, что угол между осями симметрии пи-спиральных участков примерно 120 градусов. При этом оси не пересекаются, а скрещиваются. Интересно будет посмотреть пространственную модель всей фрактальной спирали белка KAI8778976.1 Предварительный анализ показал, что фрактальная спираль имеет один излом, образуя острый угол. Возможно, что из таких субъединиц может сложиться звездчатая призма, имеющая центральный канал большого сечения и боковые каналы по числу лучей звезды.
Ранее в рассылке были опубликованы модели фрактальных спиралей белков, которые могли являться спиральными физико-химическими реакторами ультравысокого давления. На этот раз может оказаться, что существуют и звёздчатые реакторы гипервысокого давления. Это связано с тем, что центральный канал звездчатой призмы окружен более мелкими каналами, которые могут выдерживать давление первого уровня. При этом центральный канал может выдержать более высокое давление, т.к. окружён каналами высокого давления. Это мы попытаемся проверить в ближайшем будущем.
Компактная 3D графика структурного шаблона аминокислоных остатков

Компьютерная 2D модель белка 0318_KAI 8118976.1
А теперь попробуем построить пространственную модель по этой схеме вторичной структуры. Красным цветом выделены альфа-спиральные участки, синим - пи-спиральные.

Так выглядит эта фрактальная спираль, состоящая из участков альфа- и пи-спиралей. Вид вдоль оси симметрии Каждый участок альфа-спирали состоит из 4 остатков, а каждый участок пи-спирали из 10.

Та же фрактальная 4-альфа-10-пи-спираль. Вид сбоку. Виртуальный суставно-стержневой конструктор Кушелева-Дементьевой позволяет уточнять транспозиционные углы на изломах спиралей. Эта юстировка углов заменяет физическое моделирование под названием "докинг". Соседние витки фрактальной спирали в этом процессе стабилизируются межвитковыми связями, образованными радикалами аминокислотных остатков.
Пространственные модели 57 (альфа-пи) Q-спирали
Кольцегранная
Уточнение углов с помощью 3D-принтера
Модельные эксперименты на 3D-принтере

Компьютерная модель спирали Кушелева (фрактальной 75-спирали), выполненная на основе суставно-стержневого конструктора Кушелева-Дементьевой.

Параметры Q-спирали 5.9 остатков на виток и соответственно угол поворота остатка вокруг оси спирали 61 градус получились при изменении транспозиционного угла остатка с кодом 7 на минус 20 градусов.
Белок KR2. Оптогенетика. Лаборатория перспективных исследований мембранных белков МФТИ. Структура KR2 по Пикотехнологии Кушелева. Для авторов: Георг Бюлдт (Georg Bueldt) , Валентин Горделий, Валентин Борщевский, Вадим Черезов.
2017.01.02 01:54:19
Татьяна Рясина пишет:
Универсальный «переключатель» для светочувствительных клеток
https://mipt.ru/newsblog/lenta/versatil … lled_cells
"Кристаллическая структура натриевого насоса"
2017.04.22 10:57:13
Татьяна Рясина пишет:
По поиску - структура белка KR2 - находится
https://www.google.ru/search?q=%D0%B1%D … %D0%B0+KR2
Кушелев:
На уровне вторичной структуры очевидно, что нет там альфа-спиралей, заявленных в PDB.
2017.04.07 21:14:03
Татьяна Рясина пишет:
Вы могли бы сделать 2D, 3D, 4D структуру белка KR2 на обновлённой версии Пикотеха?
Кушелев: Так новая версия ещё не готова. Программист сильно занят. Пока нашёл время только на усовершенствование 2D-версии. Но даже на этом уровне хорошо видно, что РСА не "видит" даже вторичную структуру белка. Поэтому исследователи заполняют её кусками альфа-спиралей "от фонаря"
А реальная структура совсем другая.
Я конечно, могу запустить определение третичной структуры на старой версии Пикотех, но может быть много ошибок. Запускать?
2017.04.07 22:09:11
Татьяна Рясина пишет:
А самые новые апрельские структуры Вы в старой версии делаете?
Чисто житейски лучше конечно подождать версию без ошибок.
А потом выслать профессуре МФТИ.
Куешелев:
Да, пока в старой. Для спиральных участков это обычно "прокатывает", но тоже не всегда. Программные спирали бывают разные...
2017.04.08 14:13:08
Пикотехнология белков, ДНК, РНК
Татьяна Рясина пишет:
Пикотехнология тоже, как и РСА, показывает этот самый натриевый насос в белке KR2, и если да, то как?
Кушелев:
Для ответа на этот вопрос нужно исправить ошибки, т.е построить правильную 3D-модель этой белковой молекулы. Для этого нужна интерактивная версия программы Пикотех, которая уже запланирована, но будет создана не очень скоро. Программист сильно занят зарабатывание денег на жизнь, поэтому программами может заниматься раз в неделю по полчаса. Такими темпами можно год и больше писать интерактивную версию Пикотех-3D.

2017.04.07 22:08:32
Татьяна Рясина пишет:
Но конечно хочется в старой версии увидеть тоже ))))
Кушелев: Сейчас попробую сделать
2017.04.07 22:39:56
http://www.rcsb.org/pdb/explore/explore … ureId=4XTN
http://www.uniprot.org/uniprot/N0DKS8
http://www.ebi.ac.uk/ena/data/view/BAN14808
>ENA|BAN14808|BAN14808.1 Dokdonia eikasta sodium pumping rhodopsin
ATGACACAAGAACTAGGGAATGCCAATTTCGAAAATTTCATTGGAGCTACAGAAGGATTT
TCTGAAATTGCTTATCAATTTACATCACATATCCTTACGTTAGGGTACGCAGTGATGCTT
GCAGGATTACTATACTTTATCCTTACCATCAAAAATGTAGATAAAAAATTCCAAATGTCG
AACATATTATCAGCTGTGGTAATGGTATCGGCATTTTTGCTATTATATGCGCAGGCACAA
AACTGGACATCCAGTTTTACCTTTAATGAAGAAGTAGGAAGATATTTTTTAGATCCGAGT
GGTGATCTATTTAATAACGGATATCGCTATCTTAACTGGCTCATCGATGTACCTATGCTT
CTCTTTCAAATTCTATTTGTAGTAAGTTTAACTACTTCAAAATTTAGCTCTGTACGTAAC
CAATTCTGGTTTTCTGGGGCAATGATGATTATTACTGGGTACATTGGACAGTTTTATGAG
GTAAGTAACTTGACTGCCTTTTTAGTATGGGGAGCTATTTCATCTGCTTTTTTCTTCCAT
ATTTTATGGGTTATGAAGAAGGTAATTAATGAAGGAAAAGAGGGGATTTCCCCAGCAGGA
CAAAAAATACTTTCTAATATCTGGATCTTATTTTTAATATCATGGACTTTATATCCAGGA
GCTTACTTAATGCCATACCTTACTGGAGTAGACGGATTTTTATATAGTGAAGATGGCGTG
ATGGCTAGACAACTAGTATACACTATTGCAGATGTAAGTTCTAAAGTTATCTATGGTGTA
TTATTAGGTAACCTAGCAATTACATTAAGTAAAAACAAAGAGTTGGTTGAAGCAAATAGC
TAA

2017.04.07 22:39:56
Кушелев: По данным РСА - сплошные альфа-спирали, а программа Пикотех показывает пи-спирали и программные спирали. Во всей структуре попался только один виток альфа-спирали и 4 одиночных витка 310-спирали.
Понятно. Четвертичную структуру с пятеричной симметрией разглядели, а третичную структуру заполнили альфа-спиралями, которых в этом белке нет. Надо будет построить 3D модель, когда ошибки в 3D-версии Пикотех будут исправлены.
2D структура определятеся без ошибок.
2D структура с музыкальным кодом, Пикотехнология в обновлённой версии март-апрель 2017:


3D структура может содержать ошибки.
В настоящее время Пикотехнология проходит отладку в связи с обновлениями алгоритма, в несённым начиная с яваря 2017 года.

Оценка доходности
Купить книгу "Пикотехнология белков"

2017.04.08 14:22:52
Пикотехнология белков, ДНК, РНК
Татьяна Рясина пишет:
есть ли некие результаты, который можно увидеть с помощью Пикотеха по научной проблематике, которые исследователи сами увидеть не могут в силу ограниченности всей системы их методов?
Кушелев: Во-первых, можно показать им правильную вторичную структуру белков. Это поможет им сделать серию научных открытий. Во-вторых, можно дать послушать им музыку сборки белков. Это тоже может привести к серии научных открытий. Ведь на слух можно быстро определить расположение активных центров белковых молекул...
2017.04.08 20:36:10
Татьяна Рясина пишет:
по чём будут идти 3D структуры с сайта?
Кушелев:
Надо сделать дешевле, чем по старой технологии (10 000 евро), точнее, быстрее, и в более полном объёме,
т.е. не 3% структур, как даёт РСА, а 100% всех белков.
К 2023 год производительность компьютеров выросла, в день можно стало определять 10 000 структур, интернет пережил революционные изменения.
В 2023 года для внедрения Пикотехнологии актуальны публикации в западных рецензируемых журналах с указанием ссылок на программы моделирования структур белков, а не лицензирование .
Прогноз доходности на 2015 год
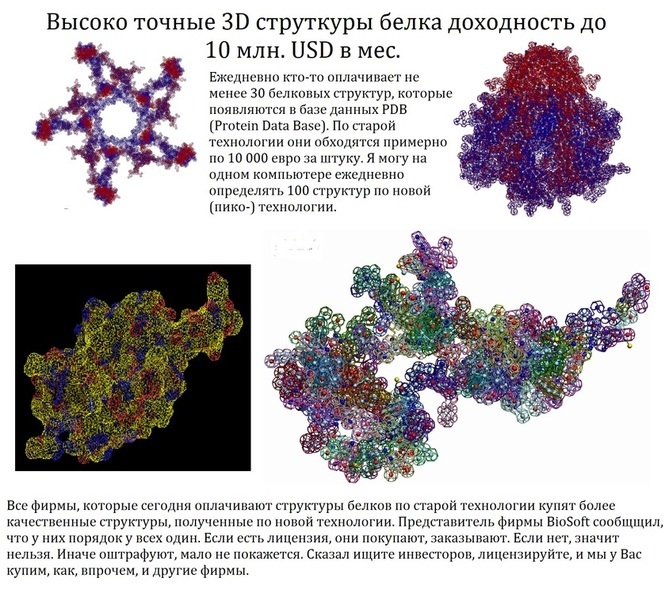
Зато потом можно будет ежедневно выполнять заказы на миллион евро и даже больше. Организовать это могут опытные менеджеры.
2016.12.16 13:22:27


В чём открытие ?
Расширенная программируемая архитектура белков
Алгоритм построения 3D структур требует уточнения композиционных углов
Композиционный генетический код, открытие 1992 года
Таблица митохондриального генетического кода и углы поворота белковых спиралей
Январь 2017. Кушелевские структемы генетического кода белкового мира и мультикомпозиционный код
2016.12.13 20:17:39
[20:12:40] Andrei: в чем открытие?
[20:12:49] Andrei: комбинация кодонов?
[20:17:14] Кушелев Александр Юрьевич: Открытие заключается в том, что три буквы кодируют не только аминокислоту, но и угол поворота этой аминокислоты относительно предыдущей:
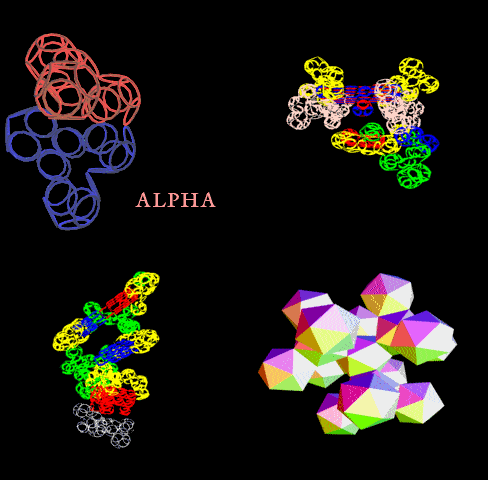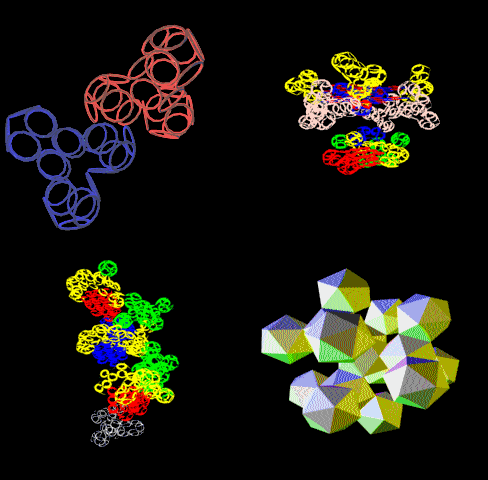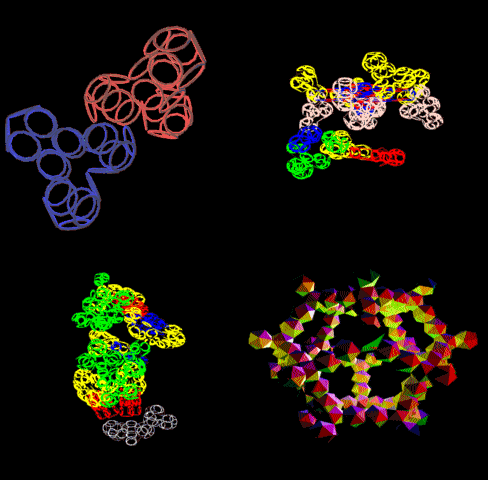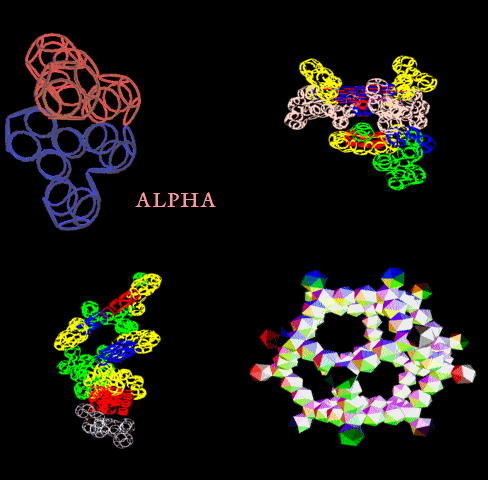
[20:17:59] Кушелев Александр Юрьевич: Этот угол (и формы аминокислот) как раз и открывают возможность определять структуры белков по таблице
[20:18:08] Andrei: а что не знали что углы есть?
[20:18:17] Кушелев Александр Юрьевич: Знали
[20:18:23] Кушелев Александр Юрьевич: Но не знали, что эти углы кодируются
Ограничения метода РСА
2017.04.05 09:13:19
Пикотехнология белков, ДНК, РНК
Татьяна Рясина пишет:
Как кратко пояснить, зачем нужен сайт с готовыми моделями?
Люди вообще слышат и видят в первый раз в жизни.
Кушелев:
Ежедневно на нашей планете кто-то оплачивает не менее 30 заказов по структурам белковых молекул. Это видно по увеличению количества белков в Protein Data Base (PDB). За каждую структуру платят в среднем 10 000 евро и ждут от нескольких недель до нескольких лет.
Структуры нужны фармацевтам, биотехнологам, генным инженерам. Потенциальных заказчиков можно найти по ключевым словам protein, nanotech
РСА (ЧЕРЕЗ ПОИСК ФОРУМА ЛАБОРАТОРИИ НАНОМИР)
Рентгенограммы РСА 2016.12.16 02:27:47
Результаты анализа рентгенограмм РСА 2016.12.16 02:37:23
97% белков не кристаллизуеся. Системные ошибки РСА 2016.12.16 02:45:32
РСА может показать чего нет и не показать что есть 2016.12.26 23:36:22
РСА цены 2016.12.21 21:15:50 , 2016.12.21 20:07:17 , 2016.12.21 20:07:17
Точность РСА 2016.12.21 17:59:55 , 2016.12.15 22:00:22 , 2016.12.15 22:00:44 , 2016.12.14 12:38:03 , 2016.12.21 14:31:08
Корреляция данных РСА с Пикотех 2016.12.18 14:41:58
РСА стремится к Пикотех 2016.12.19 15:18:09
Лауэграммы и ренгенограммы 2016.12.14 17:40:20 , 2016.12.14 18:21:50
Молекула лизоцима 2016.12.14 20:27:43
2016.12.21 21:05:30
http://www.vokrugsveta.ru/telegraph/theory/1071/

Цитата: Фотоотчет о выращивании белковых кристаллов на МКС во время 12-й экспедиции с 1 октября 2005 по 8 апреля 2006 года. Рост одного кристалла занимал 25 дней. Фото: NASA
2016.12.21 21:15:50
[20:05:27] Кушелев Александр Юрьевич: 500 000 / 120 = 4167 USD за один кристалл белка. Добавьте сюда сопутствующие работы, например, собственно РСА и анализ данных.
[21:04:47] Кушелев Александр Юрьевич: Вам эти фразы должны понравиться:

"из невразумительных точек и кругов с помощью специальных математических преобразований получают информацию о расположении атомов. Для неспециалистов выглядят как шарлатанство. Даже если решетка несложной формы, эта задача непростая, но все честно." Подробнее: http://www.vokrugsveta.ru/telegraph/theory/1071/
[21:09:54] Кушелев Александр Юрьевич: При большом везении при переборе 600 условий кристаллизации найдется 1–2 раствора, в которых появятся кристаллы. Как правило, на первом этапе они небольшие, непрозрачные и совершенно неправильной формы, то есть не подходящие для рентгеноструктурного анализа.
[21:15:41] Кушелев Александр Юрьевич: Вот ещё цитата о цене белкового кристалла: На получение одного кристалла белка ученые могут потратить десятки тысяч евро, а фармацевтические компании готовы заплатить еще большие деньги за подробную информацию о структуре какого-нибудь важного для медицины белка — «замка», к которому можно подбирать «отмычку» — лекарство.
2016.12.21 20:07:17
[19:54:52] Кушелев Александр Юрьевич: Например, узнайте цену структуры белка, кристалл которого нужно выращивать 2 года
[19:55:12] Andrei: речь не обо мне а о вас, знаете ли вы цену или просто придумываете
[19:59:00] Кушелев Александр Юрьевич: Мне сказали цену специалисты из лаборатории РСА
[19:59:15] Кушелев Александр Юрьевич: Кстати, почитайте ещё раз, как проводится анализ: http://bio.1september.ru/view_article.php?ID=200900401
[20:00:25] Andrei: Сказать это одно, а заказать другое:)
[20:01:35] Кушелев Александр Юрьевич: Вот сюда позвоните и спросите, сколько будет стоить кристаллизация белка (минимум и максимум): http://www.piboc.dvo.ru/structure/ext_l … ristal.php
[20:03:14] Andrei: Ладно, это мелочь
[20:03:31] Andrei: Надо искать кому это нужно и зачем
[20:03:46] Кушелев Александр Юрьевич: Вот Вам конкретные данные: "со ста двадцатью образцами работа еще даже не начиналась. На их восстановление потребуется не меньше полумиллиона долларов и от нескольких недель до нескольких месяцев работы." Подробнее: http://www.vokrugsveta.ru/telegraph/theory/1071/
[20:04:08] Кушелев Александр Юрьевич: 120 кристаллов белка - 500 000 долл.
[20:05:27] Кушелев Александр Юрьевич: 500 000 / 120 = 4167 USD за один кристалл белка. Добавьте сюда сопутствующие работы, например, собственно РСА и анализ данных.

























































